
Promega新型专利萤光素酶NanoLuc® Luciferase研发过程揭秘

1
研究背景
生物发光主要与定量遗传学过程有关,尽管越来越多证据证明它在细胞分析及其他方面具有相当的价值。新功能的开发促使人们寻求能够为实验方案提供更高灵敏度和可适应性的新型化学发光方法。通过天然酶固有的发光效率以及小型催化亚基在结构上与iLBPs相关的发现,使我们意识到来自深海虾Oplophorus gracilirostris的萤光素酶为我们提供了这样的机会。由于这些蛋白表现出良好的结构特性,并可以在脊椎动物和无脊椎动物中普遍表达,因此该类蛋白可为研发稳定可靠的发光报告基因提供优质选择。据报道,Oplophorus萤光素酶的底物特异性也很强,因此我们有机会设计出一种性能更为优越的改良型生物发光底物1。
为了实现这一目标,我们使用了各种诱变方案对小型催化亚基的结构优化,并与一组有机合成的新型底物类似物相结合。通过多方面的技术整合,一种新型的小萤光素酶-NanoLuc(Nluc)应运而生。这种小萤光素酶亚基只有19(kDa),能够产生极为明亮且持久的发光信号,这是其他种类萤光素酶难以企及的性能。我们通过将这些酶和底物进行工程化改造,创建了一套全新的生物发光系统,成功将哺乳动物细胞中的发光表达提高了约250万倍。

2
研究方法及结果
深海虾Oplophorus在明亮的发光云状物中分泌天然萤光素酶,这是其防御捕食的一种保护机制。经研究发现这种天然的酶结构稳定,且具有高比活度和量子产率。2它具有由两个35 kDa亚基和两个19 kDa亚基组成的异聚结构,但cDNA克隆显示生物发光活性仅与较小的亚基(Oluc-19)有关。像许多海洋萤光素酶一样,天然情况下它催化腔肠素底物产生蓝光(最大光谱454 nm),其反应不依赖ATP。
我们通过对Oluc-19氨基酸序列的分析揭示了其与细胞内脂质结合蛋白家族(iLBPs)的关联性,表明了潜在的蛋白结构对结构稳定性研究的支持。为获得最佳的酶学结构,我们进行了各种试验来探索发光底物的变体。
稳定氨基酸的预测
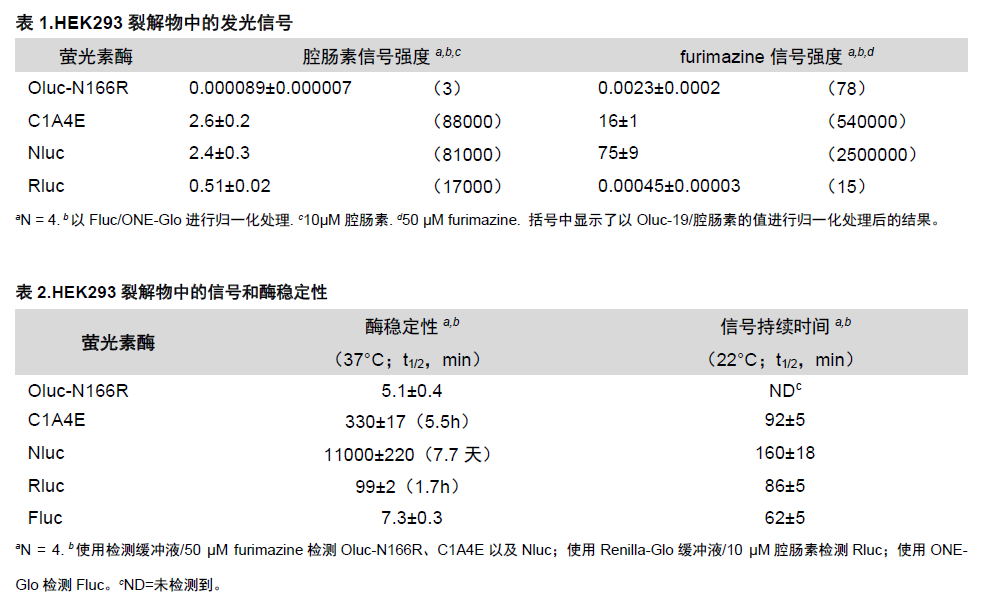
首先,3我们确定了Oluc-19与特征明确的iLBP蛋白家族之间的远距离相似度。该蛋白家族显示出高度保守的结构基序, 我们假设可以通过改变部分氨基酸突变来创建Oluc-19的更稳定变体。结果所测试的突变均产生了更高的酶稳定性,并改善了细菌裂解物中的光输出,这就进一步证实了Oluc-19与iLBPs的结构相似性。其中Oluc-N166R得到了最大程度的改善(在37°C时,其稳定性提高了约50%,发光强度提高了约3倍),并可用作定向进化的模板。
新型腔肠素底物Furimazine的结构优化和筛选
Oluc-N166R 的发光表达在大肠杆菌中分三个阶段进行了优化:
第一阶段进行一轮随机突变,以获得更明亮的发光信号。多个有益突变结合产生了C1A4E变体。在HEK293细胞裂解物中进行分析时,C1A4E的亮度约为Oluc-N166R 的29000倍(表1)。C1A4E表达量的增加与其在37°C下酶稳定性的提升相一致,其中活性保持的半衰期比Oluc-N166R延长了65倍(表2)。
第二阶段集中于优质底物的鉴定和进一步增强的发光信号的筛选。最终,我们发现与腔肠素或腔肠素h(R2=苄基)相比, Furimazine在细胞培养基中更稳定并产生更低的自发光(图1a、b)。
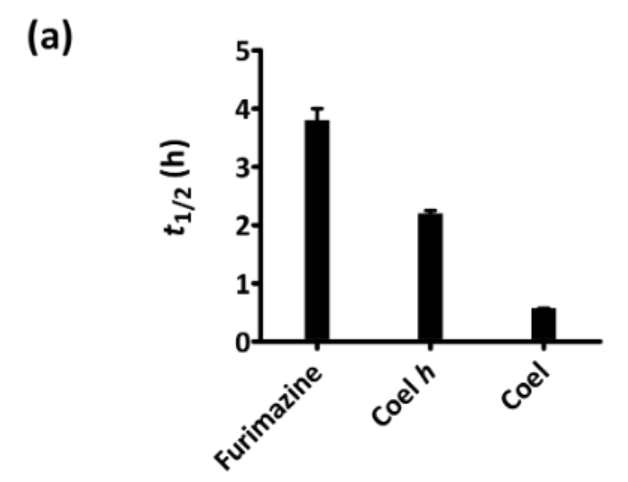
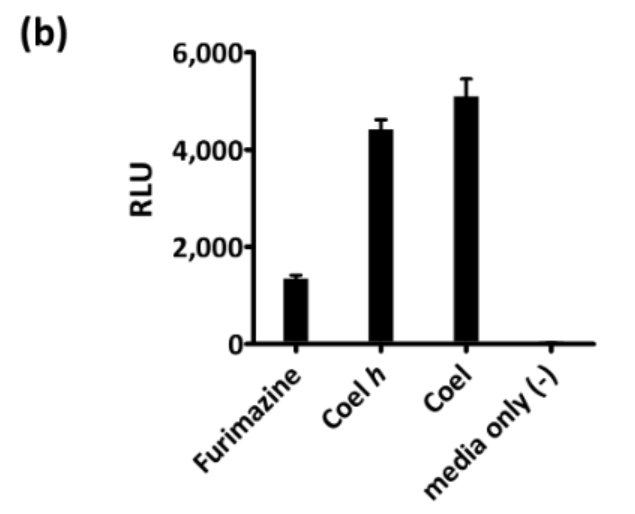
图1. (a)腔肠素,腔肠素h(R2=苄基)以及Furimazine的稳定性比较。(b)腔肠素,腔肠素h(R2=苄基)以及Furimazine的背景自发光检测。
最后阶段注重于通过Furimazine底物使发光信号最大化。使用Furimazine筛选另一个随机突变体库以获得更明亮的发光信号。结果显示:相对于腔肠素与Oluc-19组合,与Furimazine 配对的Nluc在哺乳动物细胞中产生了250万倍明亮的发光信号(表1)。
NanoLuc®表征和与Fluc和Rluc的比较
我们优化了Nluc的检测条件,以在标准实验室条件下实现高强度发光、保持信号持续时间和良好的工作稳定性(图2a,2b)。Nluc光谱图显示最大发射波长为460 nm,这与报道的野生型萤光素酶光谱一致。2相对于Rluc光谱,该光谱蓝移了20 nm,并且变窄了约20%(图2c)。因此,Nluc应该非常适合与在较长波长处有发光信号的报告基因进行叠加应用4,从而提供具有良好光谱分离的双萤光素酶检测,以支持更大的复合动态范围和灵敏度。
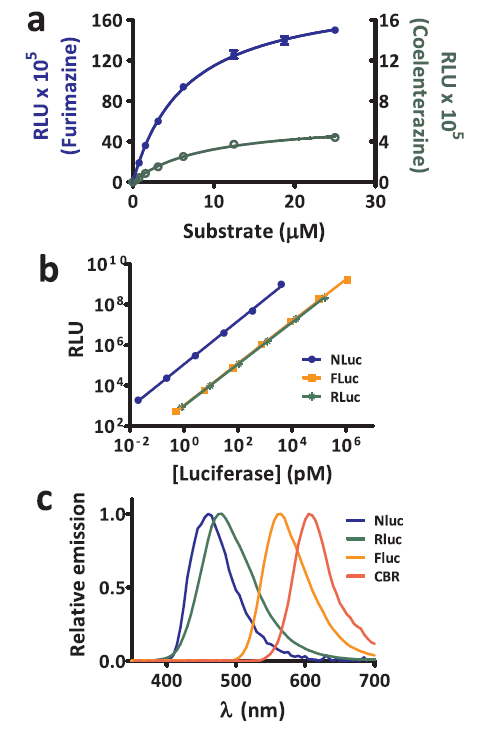
图2.(a)用Nluc滴定Furimazine和腔肠素,以确定相对信号强度和Km(n=3)。请注意,左轴和右轴具有不同的比例。(b)纯化的Nluc、Fluc和Rluc的发光强度(在10分钟时)的比较。(c)Nluc(Furimazine)、Rluc(腔肠素)、Fluc(D-萤光素)和叩头虫红色萤光素酶(CBR)(D-萤光素)的光谱图。发射波峰:Nluc(460 nm)、Rluc(480 nm)、Fluc(565 nm)和CBR(605 nm)。RLU=相对发光单位。
此外,在各种环境条件下进行比较时,发现Nluc的酶活性比Fluc更稳定。包括高温,pH值,尿素和NaCl的敏感性测定(图3)。
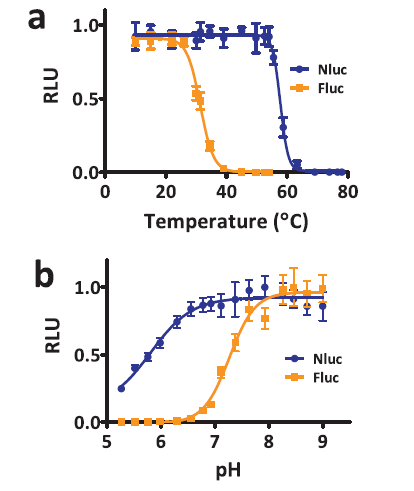
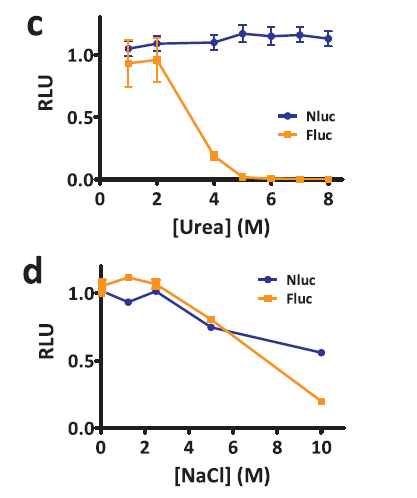
图3. 纯化的Nluc和Fluc对(a)高温(n=4)、(b)pH(n= 3)、(c)尿素(n=3)和(d)NaCl(n=3)的敏感性的比较。
作为报告基因的表现
Nluc来自海洋无脊椎动物,并且在酶学和基因上都进行了修饰,因此Nluc不太可能表现出对哺乳动物细胞特有的偏好性。免疫检测(图4a)和发光成像(图4b、c)均表明Nluc可在哺乳动物细胞中进行均匀表达,包括细胞核。此外,在表达Nluc的细胞与对照细胞之间没有明显的形态学差异。使用来自大肠杆菌和哺乳动物细胞的纯化的酶,通过质谱(LC-MS)进行总质量分析,结果表明没有翻译后修饰。两种来源的蛋白质均具有相同的分子质量,与预期的未修饰蛋白质的计算质量相匹配。
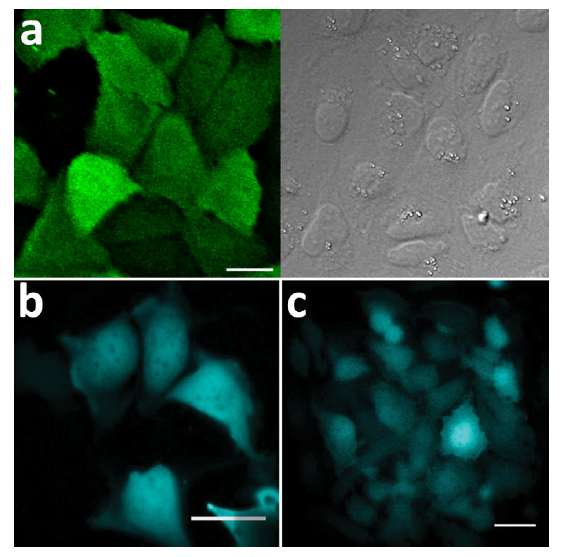
图4. Nluc在胞内的分布,通过(a)瞬时表达的 U2OS 细胞被固定并用抗 Nluc IgG/Alexa488 偶联的二级 IgG处理后共聚焦成像/ICC来确定(左图=荧光;右图= DIC);比例尺为20 μm。(b)U2OS细胞中瞬时表达的BLI;比例尺为40 μm。(c)在Hela细胞中稳定表达的BLI;比例尺为40 μm。使用单次添加Furimazine的方式在Olympus LV200生物发光成像仪上进行BLI检测。
为了获得更好的响应动力学,通常将PEST促蛋白降解序列添加至Fluc,以缩短其细胞内的寿命。5我们通过给Nluc(NlucP)进行类似的融合来评估这种方法。结果与实验预期相同,NlucP的光信号比Nluc弱,但在通路激活后显示出更快的信号增加速率和更强的总体响应(2000倍 vs.750倍)。因此可以预测的是,NlucP可以提供与转录动力学更佳的偶联,同时仍然具有良好的检测灵敏度(图5)。
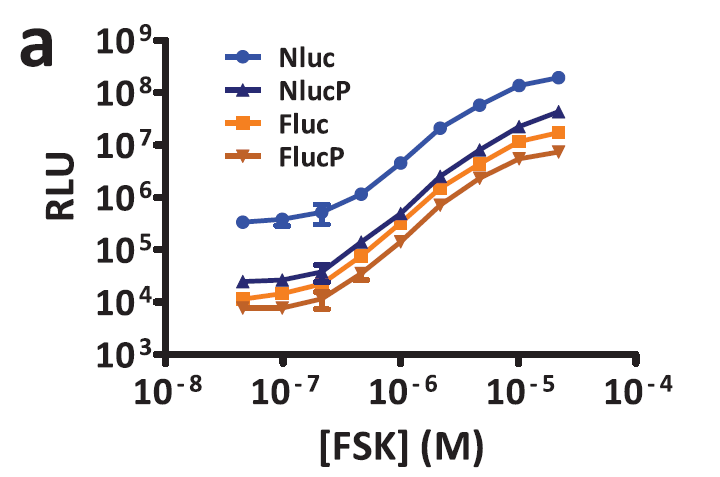
图5. (a)CRE诱导的报告基因表达。Nluc、Fluc、NlucP和FlucP接在与最小启动子连接的多个CRE下游,在HEK293细胞中瞬时表达;加入不同浓度的FSK(n=3)5小时后测得的发光信号。
作为融合蛋白标签报告基因的性能表现
Nluc以其发光信号强和分子量小等特性,成为了多种应用中的蛋白质融合标签的优先选择。6萤光素酶融合已越来越普遍地被用于检测细胞内泛素化/蛋白酶体系统(UPS)对蛋白质寿命的调控作用。
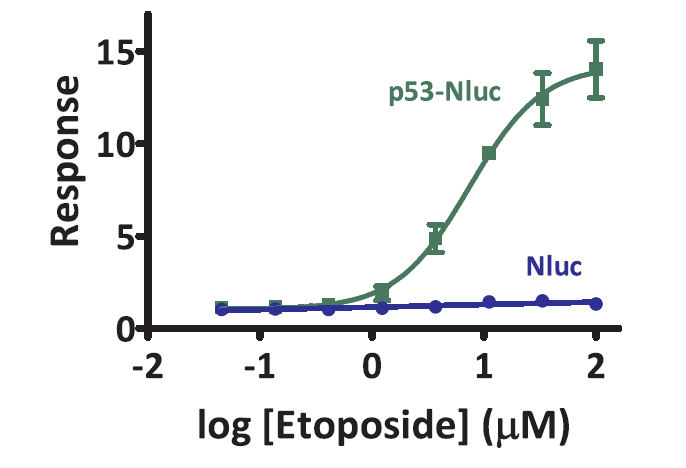
图6. 使用Nluc监测p53稳定性的调控变化。瞬时表达p53-Nluc或Nluc的HEK293细胞用依托泊苷处理6小时(n=5)。通过将处理后的样品与未处理的对照进行比较来计算应答反应值。
通过将Nluc融合到具有不同静态和动态定位模式的蛋白质的生物发光成像(BLI)方法,我们还检测了Nluc监测亚细胞定位的能力(图7)。PKCα和GR融合蛋白的表现与内源性PKCα7和GR8的亚细胞定位动力学一致,这表明Nluc并没有显著干扰融合伴侣的功能。
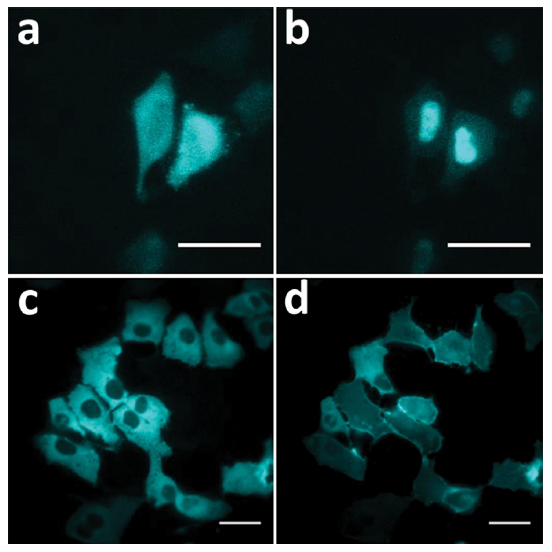
图7. 使用BLI监测Nluc融合蛋白的易位。瞬时表达Nluc-GR融合蛋白的Hela细胞显示(a)地塞米松(500 nM)处理15分钟后的胞浆定位和(b)细胞核内积累情况。瞬时表达Nluc-PKCα融合蛋白的U2OS细胞在PMA(100 nM)处理20分钟后显示(c)胞质定位和(d)质膜累积情况。比例尺为40 μm。
向培养基中添加20 μM Furimazine后立即进行生物发光成像。在这些条件下,没有明显的形态学变化(图4b、c)。暴露2小时后对细胞活力进行定量分析,同时检测表达Nluc的细胞的发光强度。综合以上结果表明,可以在活细胞中获得Nluc的高的光强度,且对细胞的正常生理影响最小。
3
总结
-
新型NanoLuc® 萤光素酶产生辉光型发光信号(信号半衰期>2h),其特异性活性比类似可进行辉光型检测的萤火虫萤光素酶(Photinus pyralis)或海肾萤光素酶的活性高约150倍。
-
在哺乳动物细胞中,NanoLuc® 未显示出翻译后修饰或亚细胞分区的迹象。
-
新型NanoLuc® 萤光素酶具有高物理稳定性,可以在高达55°C下孵育或在37°C的培养基中培养15小时以上依旧保持活性。
-
作为遗传报告基因,可以通过添加蛋白降解序列以减少细胞内累积,从而将NanoLuc® 配置为具有高灵敏度或快速应答动力学的萤光素酶。
-
添加信号序列可使NanoLuc® 分泌到培养基中且无需裂解细胞即可检测报告基因的表达。
-
将NanoLuc® 萤光素酶与其他蛋白质融合可以对其细胞内新陈代谢或定位分析进行发光法检测。
-
报告基因的定量即使在非常低的表达水平下也可以实现,以促进与内源性细胞学过程更加可靠的耦联。
4
参考文献
1. Inouye, S., and Shimomura, O. (1997) The use of Renilla luciferase, Oplophorus luciferase, and Apoaequorin as bioluminescent reporter protein in the presence of coelenterazine analogues as substrate. Biochem. Biophys. Res. Commun. 233, 349−353.
2. Shimomura, O., Masugi, T., Johnson, F. H., and Haneda, Y. (1978) Properties and reaction mechanism of the bioluminescence system of the deep-sea shrimp Oplophorus gracilirostris. Biochemistry 17, 994−998.
3. Kurowski, M. A., and Bujnicki, J. M. (2003) GeneSilico protein structure prediction meta-server. Nucleic Acids Res. 31, 3305−3307.
4. Davis, R. E., Zhang, Y. Q., Southall, N., Staudt, L. M., Austin, C.P., Inglese, J., and Auld, D. S. (2007) A cell-based assay for IκBα stabilization using a two-color dual luciferase-based sensor. Assay Drug Dev. Technol. 5, 85−103.
5. Swanson, B., Fan, F., and Wood, K. V. (2007) Enhanced response dynamics for transcription analysis using new pGL4 luciferase reporter vectors. Cell Notes 17, 3−5.
6. Simmons, S. O. (2011) Fireflies in the coalmine: luciferase technologies in next-generation toxicity testing. Comb. Chem. High Throughput Screening 14, 688−702.
7. Nishizuka, Y. (1984) The role of protein kinase C in cell surface signal transduction and tumour promotion. Nature 308, 693−698.
8. Htun, H., Barsony, J., Renyi, I., Gould, D. L., and Hager, G. L. (1996) Visualization of glucocorticoid receptor translocation and intranuclear organization in living cells with a green fluorescent protein chimera. Proc. Natl. Acad. Sci. U.S.A. 93, 4845−4850.
萤光素酶技术解决方案
双萤光素酶报告基因解决方案:
萤光素酶技术资源汇总:
如需购买相关产品请联系Promega公司:
电话:010-58256268
公司网址:www.promega.com.cn
技术支持邮箱:
chinatechserv@promega.com

产品信息

说明书查询

实验工具

技术资料









