
HiBiT蛋白标签系统
HiBiT简化了蛋白质检测,通过使用一种方便的基于生物发光的方法为检测标签化蛋白提供了一种改进的替代方法。HiBiT技术无需过表达即可检测蛋白质的动态范围,单种试剂添加更方便,还能进行活细胞检测,为研究蛋白质生物学的研究人员提供了更多的可能性。兼容灵敏的和特异性的抗HiBiT单克隆抗体,扩充了HiBiT标签蛋白的检测,使传统的基于抗体的方法可以检测HiBiT标签。
Promega HiBiT蛋白标签系统优势:
1. HiBiT标签蛋白体积小,只有11个氨基酸,对所研究蛋白影响小;
2. Promega提供多种类型HiBiT标签检测系统;
3. HiBiT检测线性范围宽,>7个数量级,可检测低至10∧(-18) 摩尔(amole)的蛋白;
4. 商品化HiBiT载体,可以快速将HiBiT标签与感兴趣蛋白构建融合蛋白载体;
5. 可应用CRISPR 技术将HiBiT标签整合到目标蛋白基因组相应位置,而无需克隆,在蛋白内源表达水平上研究;
6. 可简化传统Western blot 实验操作流程,无需抗体。
应用方向包括:表达分析,蛋白稳定性,蛋白降解,自噬,膜受体转运,病毒&细菌感染,靶细胞杀伤等。
01
如何使用HiBiT标记蛋白

通过CRISPR敲入标记内源蛋白-无需克隆
HiBiT技术通过消除插入前的分子克隆需求和简化标签蛋白的检测流程,使CRISPR介导的标签敲入成为可能。由于HiBiT标签很小,使得CRISPR插入效率高,而且生物发光使无抗体检测也具有检测内源性表达的灵敏度。
02
从5种HiBiT检测方法中选择
1.裂解型检测
在细胞裂解物,IP复合物或其他无细胞系统中对HiBiT标签蛋白进行定量检测,灵敏度高。

2.细胞外检测
定量活细胞表面或分泌型表达的蛋白。

3.印迹检测
使用简单的发光信号即可在几分钟内检测印迹膜上任意标签化蛋白。

4.活细胞细胞内检测
在细胞内表达LgBiT, 定量细胞内的HiBiT标签蛋白,无需裂解细胞。

5.基于抗体的检测
执行传统的基于抗体的检测,例如通过western blotting,免疫组织化学,pull-down的方法检测HiBiT标签蛋白。

03
HiBiT技术应用举例
监测受体内化:

HiBiT检测方法消除了受体内化研究中基于抗体检测的必要。使用HiBiT技术,操作流程中去除了多个抗体结合步骤和相关的洗涤步骤。只需加入检测试剂,即可测量出发光信号。
定量蛋白丰度和降解:
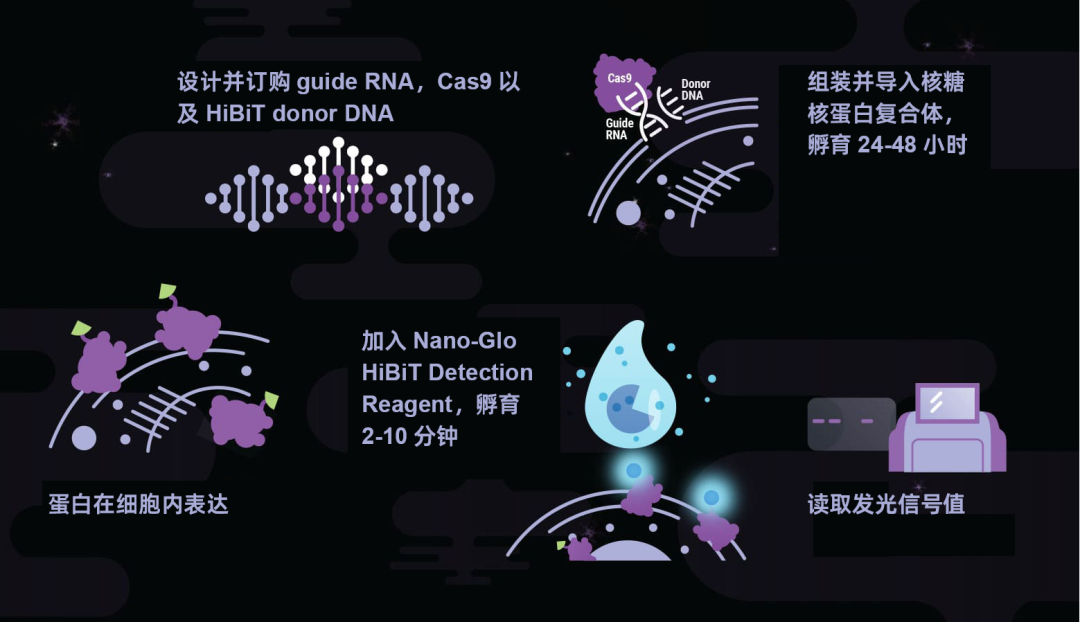
经典的表位标记方法在通量或灵敏度上是有限的,要求使用高质量的抗体,但也可能只产生半定量的结果。HiBiT标签将生物发光的简便性和灵敏度引入到蛋白质丰度的研究中,可对内源性水平的蛋白质进行定量,即使是那些维持在低表达水平的蛋白质。
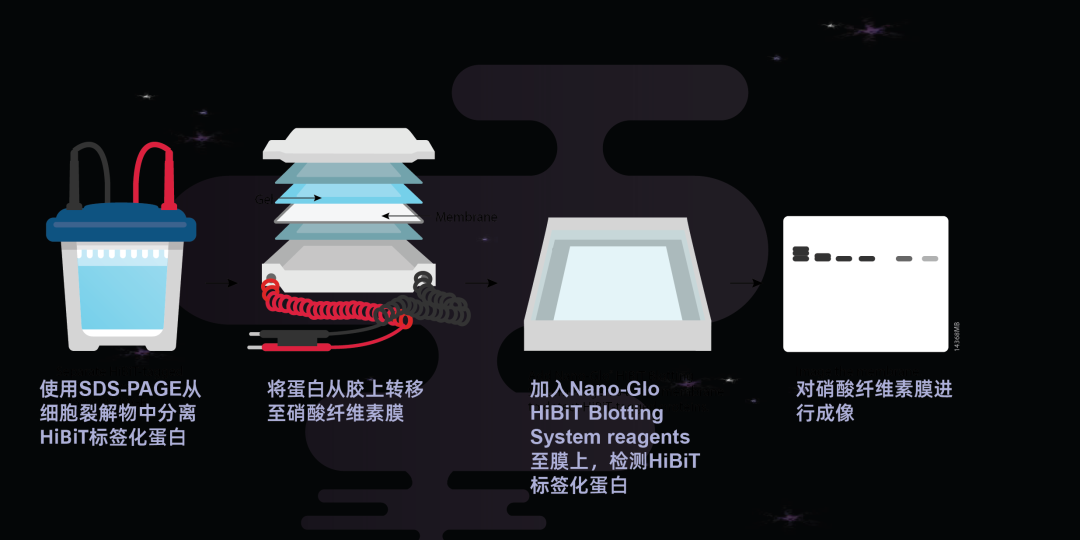
快速简单的免疫印迹:

HiBiT印迹系统是一种灵敏、快速的检测方法,可以替代费时费力的免疫印迹技术,且无需抗体。
“
1. 什么是HiBiT印迹检测?
-
HiBiT印迹检测:一种快速、灵敏的Western Blot替代方法
-
5分钟内出结果
-
无需抗体
-
背景低,灵敏度高
-
无需洗涤或封闭步骤
HiBiT是一种基于蛋白质互补的生物发光检测方法。HiBiT标签被添加到你感兴趣的蛋白质中,当互补的LgBiT亚基与HiBiT结合时,形成一个具有功能性的萤光素酶进行检测。
“
2. HiBiT与传统的表位标签不同
-
在传统的Western blotting中,你需要一个目标蛋白或表位标签的抗体,以及一个检测该种抗体的方法。
-
使用HiBiT,无需抗体,消除了实验处理步骤,并最大限度地缩短了手动操作的时间。
“
3. HiBiT蛋白标签的优势
-
易于定量及检测
-
无需封闭
-
与基于抗体的方法不同,没有背景,使用HiBiT印迹,检测试剂是否非特异性地结合到膜上并不重要,只有当它与带有HiBiT标记的蛋白质结合时,才会有发光信号。
“
4. 如何应用HiBiT标签
-
监测细胞内的蛋白丰度
-
蛋白降解研究
-
监测受体内化
-
插入并检测CRISPR敲入效率
“
5. 参考文献
1)Nakashoji, A. et al. (2020) Identification of a Modified HOXB9 mRNA in Breast Cancer. Journal of Oncology. Article ID 6065736,
2) Schwinn, M.K. et al. (2020) A Simple and Scalable Strategy for Analysis of Endogenous Protein Dynamics. Sci. Rep. 10(1), 8953.
3) Tange, N. et al. (2020) Staurosporine and venetoclax induce the caspase-dependent proteolysis of MEF2D-fusion proteins and apoptosis in MEF2D-fusion (+) ALL cells. Biomed Pharmacother. 128, 110330.
4) Ranawakage D.C. et al.(2019) HiBiT-qIP, HiBiT-based quantitative immunoprecipitation, facilitates the determination of antibody affinity under immunoprecipitation conditions. Sci. Rep. 9(1) 6895.
5) Sasak, M. et al. (2018) Development of a rapid and quantitative method for the analysis of viral entry and release using a NanoLuc luciferase complementation assay. Virus Res. 243, 69-74.
6) Tamura, T. et al. (2019) In Vivo Dynamics of Reporter Flaviviridae Viruses. J Virol. 93(22), e01191-19.
相关阅读
技术 | CRISPR / Cas9 HiBiT敲入:研究内源性蛋白质动力学的可扩展型方法

产品信息

说明书查询

实验工具

技术资料












