
Promega萤光素酶技术有声故事
Podcasts for Luciferase Technologies
2022
寻找好声音活动持续进行中,
欢迎大家踊跃报名!
我们等着你,来实现主播梦!

START

使用NanoBRET™和NanoBiT®检测研究
Myc和Max蛋白相互作用
|
麦主播:发光发亮 |
|
|
大家好,我是麦主播发光发亮,字正腔圆,雄浑有力,给你一种醒神的效果哦! |
|
Story Introduction
故事内容简介
研究背景
研究方法
研究结果
资源列表
我们使用Promega公司生产的NanoBRET™检测,NanoLuc®萤光素酶(NanoLuc®)和HaloTag®配体标记的相应蛋白,对Myc-Max蛋白相互作用(PPI)抑制剂进行了鉴别。此外,我们还使用Myc-Max NanoBiT®互补检测对特定Myc-Max PPI进行了确认。通过Myc-Max NanoBRET™检测,我们对约40000种化合物进行了高通量(HTS)筛选(384孔板检测模式),从而鉴别相关PPI抑制剂。我们对剂量反应实验以及反向筛选实验鉴别的活性化合物继续进行研究,从而确定了可破坏Myc-Max异源二聚化的新化合物。
Myc蛋白家族对细胞结局具有多种调节作用。由于存在遗传和表观遗传学突变,因此多种人体肿瘤中Myc基因均已失调(1,2)。Myc与其活化配偶体Max发生异源二聚化后,Myc方可发挥其大部分生物活性(3,4)。通过小分子化合物抑制Myc和Max之间的蛋白相互作用是抑制Myc活性的一种合理方法(2-4)。在本研究中,我们评估了两种Promega公司开发的,基于NanoLuc®萤光素酶技术的细胞水平Myc-Max 蛋白相互作用(PPI)研究的检测系统。基于BRET技术的(生物发光共振能量转移)检测系统(NanoBRET™)和NanoBiT® (NanoLuc® Binary Technology)检测系统(结构互补报告基因检测系统)均可用于化合物高通量筛选,从而鉴别PPI抑制剂。在本文中,我们介绍了两种检测系统优化结果以及后续筛选(用于鉴别抑制Myc-Max异源二聚化的新化合物)结果。
使用NanoBRET™检测系统时,我们选择了MCF10A细胞系(ATCC目录号:CRL-10317),因为该细胞系中内源性c-Myc水平较低且适合高通量筛选(HTS)。根据Promega公司以HEK293细胞为例开发的标准操作规程,我们设计融合蛋白构建体,并将其转染至细胞中,然后对其表达水平进行检测。图1为NanoBRET™ PPI检测的示意图。针对该检测,我们测试了Myc和Max蛋白氨基和羧基端的多种融合标签定位方式的融合蛋白构建体。我们另外使用模拟供体构建体(由Promega提供)(不含受试蛋白)查看受试化合物对BRET的非特异性抑制作用。此外,在检测优化期间,我们对不同转染比例融合质粒进行了测试,结果表明受体与供体的比例在100:1时为最佳。NanoLuc®底物和HaloTag®配体(目录号:N1663)均购Promega公司。NanoBRET™信号测量仪器为PHERAstar FSX微孔板读数仪(BMG Labtech),通过滤波器分离供体和受体信号。NanoBiT®互补检测试剂购自Promega公司,为定制合成的NanoBiT®大亚基(LgBiT)和小亚基(SmBiT)分别与Myc和Max融合表达的载体。使用Nano-Glo®活细胞试剂(目录号:N2013)测量LgBiT和SmBiT亚基的相互作用。数据分析软件为Genedata Screener®(13版;Genedata AG)。以NanoBRET™受体/供体信号比的形式报告NanoBRET™的结果。以相对发光信号的形式报告NanoBiT®的结果。
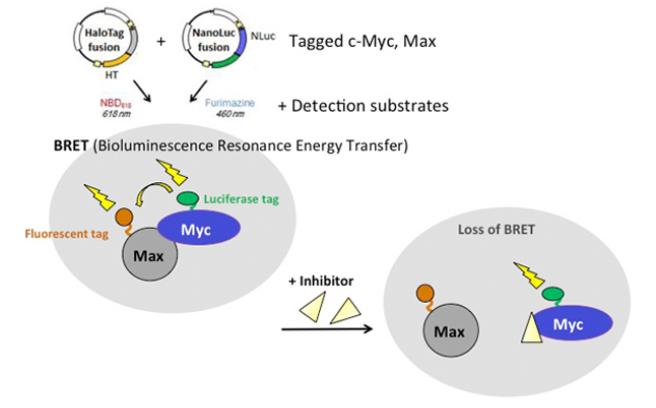
图1. NanoBRET™ PPI检测的示意图,其中NanoLuc®萤光素酶标记的Myc为供体,HaloTag®-Max融合蛋白为能量转移受体。具有任一标签的Myc和Max融合蛋白(被用于检测的确认。筛选时,我们使用了Max-HaloTag®和Myc-NanoLuc®构建体。
1. 筛选确认
由于缺乏可用作工具化合物的有效适合化合物,我们将未经HaloTag®配体处理的细胞用作NanoBRET™检测的阴性对照,进行了检测确认。首先,我们利用棋盘测试对检测方法的变异性进行了测试。Z'值≥0.5,表明检测方法重现性良好(图2)。

图2. 棋盘测试(有或无HaloTag®配体)。在150 mL组织培养物处理后的培养皿中,用蛋白质融合构建体瞬时转染MCF10A细胞并过夜培养。取转染后细胞对384孔检测平板(Corning,目录号:3570)进行铺板。细胞铺板5小时后,根据所有微孔NanoLuc®萤光素酶和HaloTag®信号原始值的对应值计算NanoBRET™比值。使用Genedata Screener®(Genedata AG)将该比值(有或无HaloTag®配体)分别标准化为100%和0%。
2. 筛选库
首先,我们对一系列具有生物活性的化合物(商业和内部化合物保藏库)进行了测试。如图3所示, 设定PPI抑制剂活性HIT率临界值为80%,HIT率约为1.5%。如图3所示,可能存在荧光化合物,从而导致受体通道信号升高,进而人为导致NanoBRET™比值升高。由于初步结果较好,因此我们还筛选了其他化合物库。通过此次筛选,我们选择了部分“活性化合物”或“HIT化合物”,并采用图4所示的相同检测方法开展了剂量反应实验,从而证明Myc-Max检测方法的抑制具有剂量依赖性。
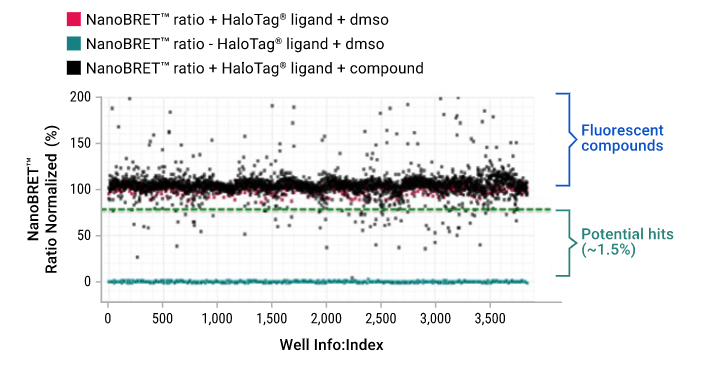
图3. 使用一系列具有生物活性的化合物筛选NanoBRET™检测的化合物。单份筛选时,将溶于DMSO的化合物原液加至检测培养基(Lonza,目录号:CC-3153,不含酚红和生长因子)中,从而使化合物终浓度为20 µM。根据NanoLuc®和HaloTag®信号原始值计算NanoBRET™ 比值。使用Genedata Screener®(Genedata AG)将该比值(有或无HaloTag®配体)分别标准化为100%和0%。化合物HIT临界值为≤80%,仅DMSO组NanoBRET™ PPI活性为100%。如果微孔对应活性大于100% NanoBRET™比值,则表明某些化合物可能造成荧光伪影信号。
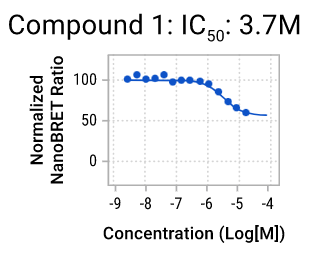
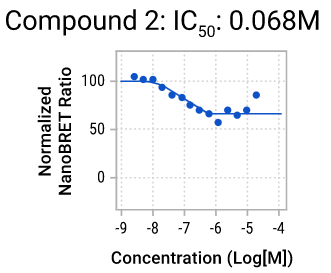
图4. Myc-Max NanoBRET™筛选实验中活性化合物的剂量反应数据。我们在14个浓度水平剂量反应实验(使用Myc和Max融合蛋白构建体瞬时转染MCF10A细胞)对初步筛选中可降低NanoBRET™比值的化合物进行了重新测试。根据NanoLuc® 和 HaloTag®信号原始值计算NanoBRET™比值。使用Genedata Screener®将该比值(有或无HaloTag® 配体)分别标准化为100%和0%。图中列出了化合物1和化合物2的IC50值。
3. NanoLuc®反向筛选检测
我们观察到部分化合物可降低供体萤光素酶信号,而且信号降低程度大于 NanoBRET™ 阳性样品。因此,我们迫切希望探明所选化合物是否对供体信号具有抑制作用,从而导致受体信号降低,进而导致NanoBRET™比值降低。这有助于避免假阳性结果。如图5所示,当无HaloTag®配体时,化合物可导致发光信号降低,表明化合物对NanoLuc®萤光素酶具有一定影响,而不是Myc和Max蛋白相互作用。在NanoBRET™ 检测中,最佳特异性PPI 抑制剂为化合物3。
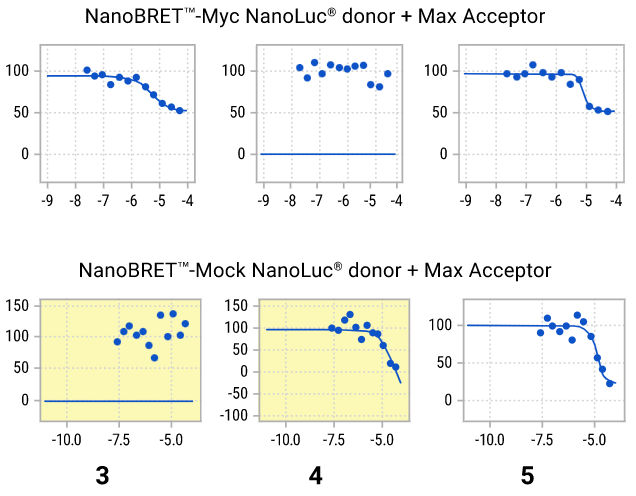
图5. 使用Myc或模拟供体进行NanoBRET™ 化合物筛选和反向筛选。Myc和Max融合蛋白表达于MCF10A细胞中。此外,我们将模拟 NanoLuc® 供体(购自Promega公司)以及Max受体融合蛋白表达于细胞中,从而确定化合物是否对检测数据具有非特异性影响。化合物3的结果较为理想,该化合物特异性较好,对NanoLuc®信号无剂量依赖的抑制作用。结果表明,化合物4和化合物5均可降低NanoLuc®信号。在NanoBRET™筛选实验中,化合物4无活性,但化合物5可降低NanoBRET™比值。X轴的值为化合物浓度(log10 [M]),y轴的值为标准化的NanoBRET™比值。
4. NanoBiT®互补检测
据相关文献报告,可采用酶互补检测方法对Myc-Max异源二聚体PPI进行评价(5)。因此,我们采用NanoBiT®互补检测方法对蛋白相互作用进行了确认。如图6所示,当两个NanoBiT®亚基与Myc和Max蛋白(滴定的配偶体为0水平)融合时,我们观察到互补现象。然后,我们测试了是否可通过NanoBRET™检测用HaloTag®配体共表达一系列Myc或Max蛋白,从而破坏互补作用。如图6所示,我们观察到Myc或Max蛋白与各自配偶体存在竞争作用且呈剂量依赖性,表明可检测到异源二聚化的破坏作用。
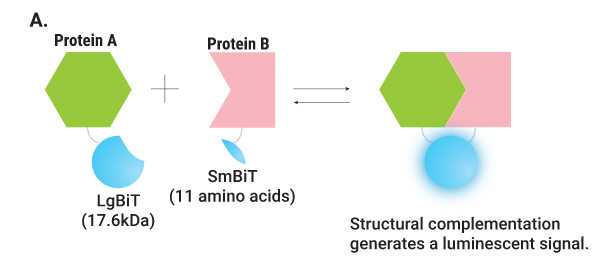
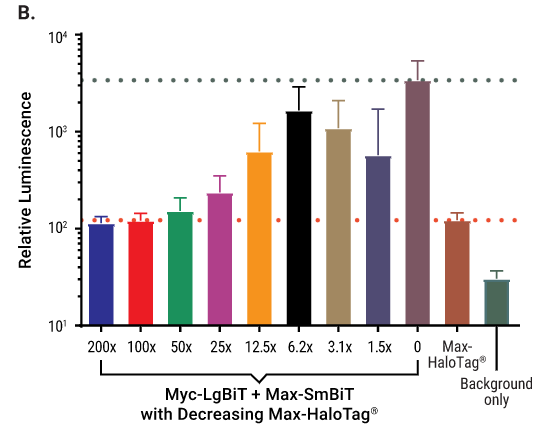
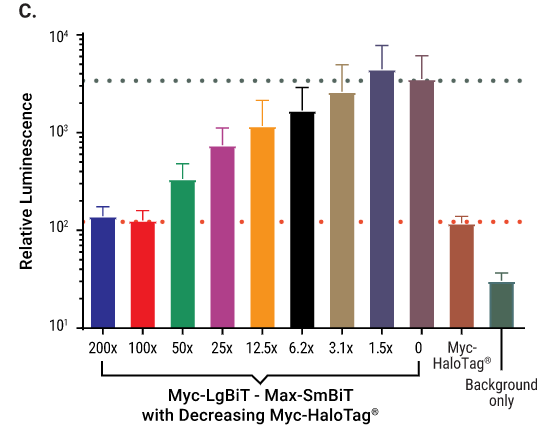
图6. NanoBiT®互补检测概述以及与Max-和Myc- HaloTag®融合蛋白表达构建体(共表达Myc和Max NanoBiT®载体)的竞争。图A. 蛋白质A和B分别与LgBiT和SmBiT融合并于细胞中表达。两种蛋白发生相互作用,从而导致LgBiT和SmBiT发生结构互补并产生发光信号。对于表达Max(图B)和Myc(图C)HaloTag®融合蛋白表达构建体,将规定比例的质粒DNA量转染至HEK293细胞中(装于100 mL组织培养皿中)。细胞转染约20小时后,使用转染后的细胞重新对384孔板(Corning,目录号:3570)进行铺板并执行NanoBiT®互补检测。本底指从加入细胞但未加入NanoLuc®底物的微孔测得的发光信号值。测量发光信号前,其他微孔均应加入NanoLuc®底物。
5. NanoBiT®互补反向筛选检测
为确定NanoBiT® Myc/Max 检测方法的特异性,我们采用不相关的p53-Mdm2 NanoBiT®检测作为反向筛选方法。如图7所示,Nutlin-3为p53-Mdm2相互作用的特异性抑制剂,可破坏本检测中p53和Mdm2之间PPI。我们还在表达Myc-和Max-NanoBiT®构建体的细胞中对nutlin-3(不会破坏这两种蛋白伙伴的互补作用)进行了测试。我们平行使用两种检测方法进行了检测,从而对潜在Myc-Max PPI抑制剂的特异性进行测试。
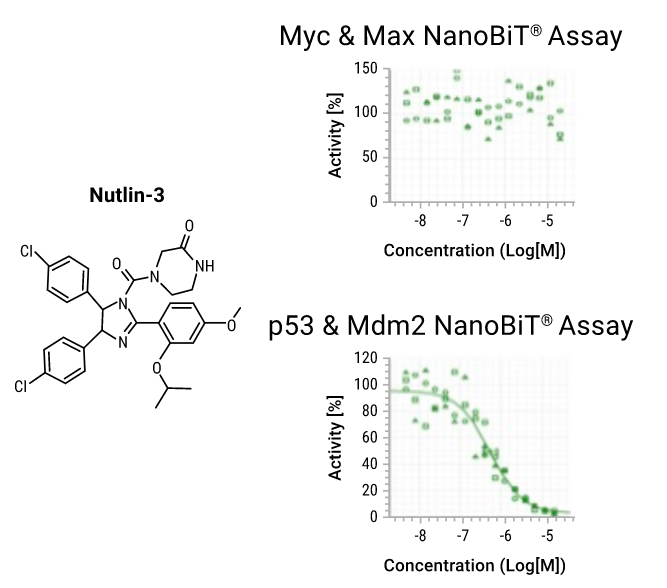
图7. NanoBiT®检测中Myc-Max PPI筛选的反向筛选。我们使用相应融合蛋白对在HEK293细胞中进行表达,并在剂量反应实验中对这些细胞中的nutlin-3(一种p53-Mdm2相互作用PPI抑制剂)(Sigma,目录号:N6287))活性进行了研究。
NanoBRET™检测和 NanoBiT®互补检测均为同类方法,适用于活细胞中PPI高通量筛选平台。目前存在多种用于细胞中PPI研究的有效工具,可与现有生化方法互补。两种基于NanoLuc®萤光素酶的检测系统的操作均较为简便,且稳健性和灵敏度均较好。可结合使用两个系统对非特异性和假阳性信号进行反向筛选。
参考文献:
1. Meyer N., Penn L.Z. (2008) Reflecting on 25 years with MYC. Nat. Rev. Cancer 8, 976–90.
2. Conacci-Sorrell M. et al. (2014) An overview of MYC and its interactome. Cold Spring Harb. Perspect. Med. 4, a014357.
3. Jung K.Y. et al. (2015) Perturbation of the c-Myc-Max protein-protein interaction via synthetic α-helix mimetics.J. Med. Chem. 58(7),3002–24.
4. McKeown, M.R. and Bradner, J.E. (2014) Therapeutic Strategies to Inhibit MYC. Cold Spring Harb. Perspect. Med. 4,a014266
5. Raffeiner P. et al. (2014) In vivo quantification and perturbation of Myc-Max interactions and the impact on oncogenic potential. Oncotarget 5(19), 8869–78
相关产品:
NanoBRET™ Transcriptional Protein Assays(#N1870)
NanoBiT® PPI MCS Starter Systems(#2014)
NanoBiT® PPI Flexi® Starter Systems(#2015)
Nano-Glo® Live Cell Assay System(#N2013)
技术资料:

点击图片进入萤光素酶技术资源页面

NEXT EPISODE
下期节目预告
节目名称:冠状病毒研究中萤光素酶标记的报告基因假病毒颗粒的测量选择
播出时间:2022年4月
麦主播:白小白
麦电台寻找好声音
Finding The Nice Voice

加入麦电台,让更多人听到你的声音。


|
|
萤光素酶有声故事汇 萤光素酶生物发光技术从自然界到实验台,每一步的变化都有着无数的故事和科学家的努力。跟随我们的脚步,慢慢聆听生物发光技术的前世今生和最新的技术进展。 |
|
|
萤光素酶技术讲座合集 萤光素酶技术系列讲座为大家循序渐进,由浅入深,逐步介绍了生物发光机理,萤光素酶基础知识及技术应用。 |
|
|
萤光素酶技术Glo学院小程序 这是一个不仅可以储备知识,同时还能赢学分兑好礼的平台。在这里,我们整合了关于萤光素酶基本知识和技术应用的优质资源,从文章解析到视频讲座,从线上直播到线下活动,从产品指南到技术答疑等多个方面为大家呈现。 |
|
|
点击了解更多萤光素酶技术应用与资源 |


产品信息

说明书查询

实验工具

技术资料











